|
|||
|
|||
|
trimAl はアライメント困難な領域を自動的に削除するプログラムです.Mac,Windows の両方で解析が可能です.html 形式のファイルが出力され,アライメント困難な領域が一目で分かるのも良いです. |
|||
 |
|||
|
|||
こちらの Commandline version の downloads をクリックして,pc に合わせたインストーラーをダウンロードします.trimAl/source に cd で入って,make と入力してください (ダウンロードして得られる README を参照). |
|||
|
|||
trimalTEST.tar.gz |
|||
|
|||
| マニュアルはこちらにあります.オプションについてはこちらにまとめられています. trimAl/dataset/example1.phy というファイリップ形式のアライメントを例に説明します.この .phy ファイルはプログラムをダウンロードすると同時に得られます.なお trmAl はfasta や nexus などのファイルも読み込むことができます. コンパイルしてできた trimal を dataset フォルダにコピーしてください.その後,dataset フォルダに cd で移動して,
と入力すると,output1 に結果が出ています.削除した部分を output1.html をダブルクリックすることによってブラウザで見ることができます.
-gt 0.8 によって,20% 以上ギャップがあるか,あるいは similarity score が 0.001 よりも小さいサイトを削除します.
より細かい設定は,こちらをご覧下さい.オプションについてはこちらにまとめられています.
|
|||
|
|||
|
|||
|
|||
 |
|||
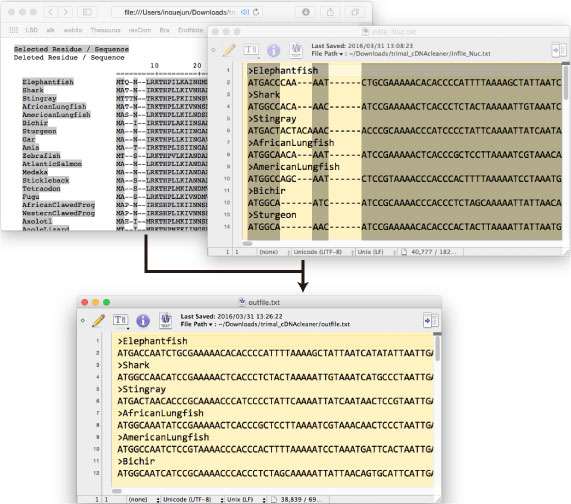
| Perl: trimal_cDNAcleaner.tar.gz Python: trimal_cDNAcleanerPYTHON.tar.gz: v1.5 に対応しました(2024 年 12 月)。 |
|||
|
|||
 |
|||
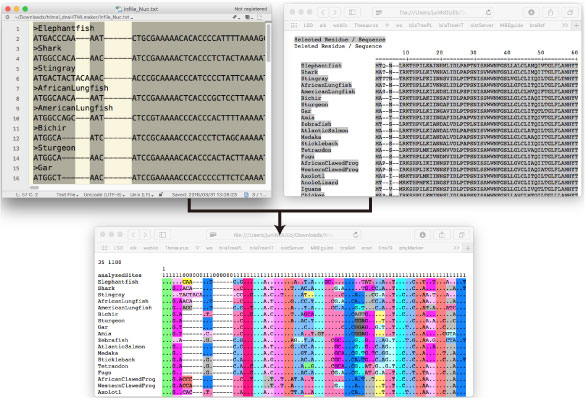
trimal_dnaHTMLmaker.tar.gz (2017 年 11 月) |
|||
|
|||
 |
|||
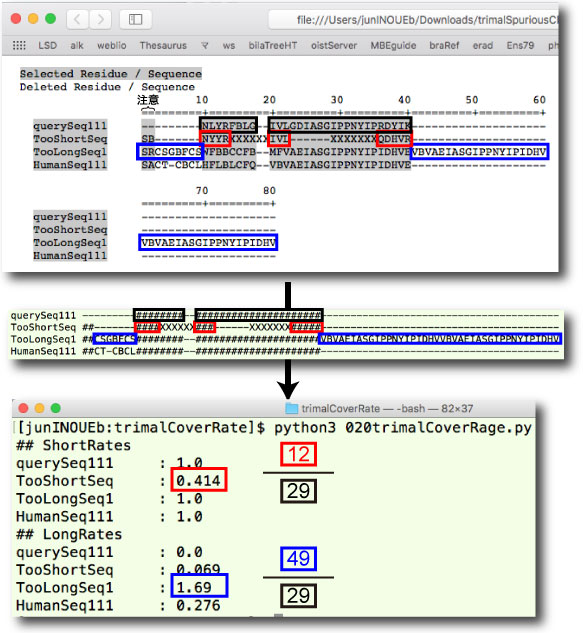
| trimalCoverRate.tar.gz trimalCoverRatePERL.tar.gz (2017 年 12 月) |
|||
|
|||
シーケンスファイルの最後の配列が,リターンで終わっていない場合に,このメッセージが出ました.配列の最後は,必ずリターンを入れておきましょう (2013 年 9 月). |
|||