|
|||
| 2014 年 12 月 13日 改訂 | |||
|
ヒトゲノム解析センター (HGC) が提供している SuperComputer に関するメモです.HGC の SuperComputer は,使用料金を払えば誰でも使うことができます.月極で支払い可能です.個人で使う場合は,大学に所属している方は一ヶ月 5000 円程度です.詳細はこちらです. |
|||
|
|||
アレイジョブについて調べる場合,以下のように検索すると良いです.
"SGE_TASK_ID" は SGD のシステムで使われているユニークなキーワードです.アンダーバーとハイフンは google 検索で無視されるので,クオーテーションマークで囲んでいます. |
|||
|
|||
tar の解凍は遅い
|
|||
|
|||
ログイン
または、
としてログインします.その後パスワードを聞かれるので答えます.「logout」あるいは「exit」でログアウトします.
でログインしているノードを確認します.gw 系ノードは解析向きではないので,
によって遊休しているログインに入って解析を行います. |
|||
|
|||
test.sh という名前のファイルを作成し,以下を保存します.「qsub test.sh」でジョブを投入します.job ファイルは名前の先頭が数字だと,qsub しても受け付けてもらえません.
|
|||
|
|||
job の投入
上記だと Home directory にアウトファイルができます. qsub したディレクトリにアウトファイルを保存
投入した job の状況
job ID を指定して job を削除
UID を指定して job を削除
ログインしているノードを確認
指定した [jobid] の詳細情報を確認
ファイルの数をカウント
ディレクトリの数をカウント
|
|||
| qlogin | |||
qlogin は解析を始める前に qlogin によって,解析用のノードに入るコマンドです.ただし,HGC に電話して聞いたところ,qlogin はどうもアレイジョブのときに行う操作のようです.qsub で解析を行う場合は,qlogin しないで gw ノードから直接 job を投げても良いみたいです (2010 年 10 月). SGE にバッチジョブを投入する前には,メモリ使用量の確認を行います.job を事前にテストする場合は,ログイン時に入った gw ノードから,
と入力し,intr.q にログインします.これで遊休しているノードにログインできます.
でノードを確認します.このとき jobID
を覚えておきます.バッチファイルで実行する job をコマンドラインに直接入力して走らせます.
10 秒ぐらいで解析が終わります.結果がスクリーンアウトされたら,exit でログアウトします.
で利用した最大か総メモリサイズを確認します.20 秒ぐらい時間がかかります.881 M かかるという結果を得ました. |
|||
| RAxML の解析 | |||
(最近 100 以上の RAxML 解析は,以下のコラムにあるアレイジョブで走らせています.大量のジョブを走らせる場合は,アレイジョブの方が楽ですし,クラスタにも問題が生じにくいです.OIST のテクニシャンの方に見ていただいたので,信頼性が高いはずです [2014 年 12 月])
job は 5000 までしか受け付けてくれません.それ以上になると,以下のようなメッセージが出ます.
アウトファイルの処理
ユーザーごとに使用できるディスク容量は限られているので,解析が終わったファイルは削除した方が解析がスムーズに進むような気がします. ############################## RAxML のコンパイル
raxmlHPC を得ようとすると,以下のエラーメッセージが出ました.
ちなみに,Pthread version (raxmlHPC-PTHREADS-SSE3) では -T2 が最も速かったです.-T 6 はなかなか解析が終わらないので途中で辞めました.
|
|||
|
|||
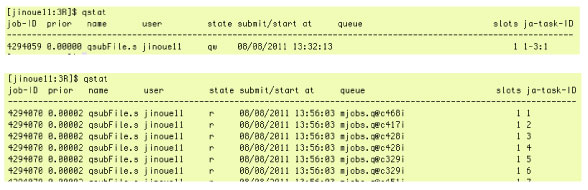
数千ぐらいのジョブは,アレイジョブとして投入します.アレイジョブは最大 75,000 タスクまでです.こちらをご覧下さい.アレイジョブを行う場合は,以下に説明のある qlogin によって計算用のノードに入って,一つのジョブに必要とされるメモリを確認する必要があります. アレイジョブがうまく走ると,「qstat」によって以下の待ち受け画面 (state: qw) と解析画面 (state: r) が得られます.state: qw では,一番右の「1-3:1」が 3 つの job をアレイジョブによって行うことを示します.state: r では 26 個の job が同時に解析されていて,その 6 番目までの状態を表示しています. |
|||
 |
|||
RAxML 解析
|
|||
| その他 | |||
| ファイル数を表示 ディレクトリ内部のファイル数を表示します.
|
|||
| リンク | |||
HGC の supercomputer 利用に関するリンクです.利用者 ID とパスワードがないと入れないページもあります. Webmail スパコン利用案内 |
|||
|
|