|
|
2020 年 3 月 28 日 井上 潤,米澤 隆弘
|
このサイトでは dN/dS 値を推定することで,タンパク質コーディング遺伝子 (DNA 配列) に働いた正の自然選択を検出する解析 (dN/dS 検定) を紹介します.PAML に含まれるプログラム codeml を使います.バージョンは paml4.7a を使っています.
用語説明
dN: 非同義置換率 (非同義座位あたりの非同義置換数)
dS: 同義置換率
(同義座位あたりの同義置換数)
ω: ω= dN/dS (ω はオメガと読みます)
dN/dS < 1 なら負の自然淘汰 (機能的制約) が働き,dN/dS = 1 なら中立的な進化状態 (機能的制約のゆるみ) にあり,dN/dS > 1 なら正の自然選択 (positive selection; 適応的な進化),が働いている (いた) と推測します (松井ら,2008).
全体像
codeml では,枝モデル (branch models), サイトモデル (site models), 枝サイトモデル (branch-site models) の 3 種類の解析が可能です.ここでは,
1) 対立仮説と帰無仮説の両方で codeml による計算 (枝,サイト,枝サイトの各モデル) によって ω を推定し,
2) 得られた尤度に基づいて、Excel を用いて尤度比検定を行い、有意な結果が得られたか確認する,
という二段階の作業を紹介します.
例題として,Yang (1998) の "small data set" を使っています.以下をダウンロードしてください (examples/lysozyme とほぼ同じファイルです).codeml のコンパイルはこちらを参照してください.
lysozymeSmall.tar.gz
このサイトは,2014 年 3 月末に米澤さんから教えていただいた dN/dS 検定の方法と結果の解釈を,井上がまとめたものです.米澤さんはこの解析手法を熟知していますが,井上は dN/dS 検定を論文に用いたこともないので,充分な理解をしていません.私の不勉強のために,文章におかしな部分があるかも知れません.
注意: paml4.9a では解析が動きませんでした.paml4.8a は動きます (こちらの例題に入っています).
|
|
|
枝モデルは,遺伝子系統樹のある枝で正の自然選択が働いたかどうかを検定する解析モデルです (Yang 2009, P258).
lysozymeSmall ディレクトリにターミナルで移動してください.コンパイルして得られた codeml を同じディレクトリにコピー&ペーストしてください.枝モデルはサイトごとの変異パターンは考慮しません.このため,codeml.ctl ファイルの NSsites (サイト間で ω を変化させるオプション) を
NSsites = 0
として,ω をサイト間で変化させずに解析を行います.
一種類の ω を推定する場合
まず,対立仮説 (特定の枝で ω の値が異なる) のもとで解析を行い,結果を mlcB2 という名前のファイルに保存します.
codeml.ctl ファイル: パラメータを以下に設定してください.
outfile = mlcB2
model = 2
model オプションは,ω の変動を枝間で調節します.
0: すべての枝で ω が均一.
1: free ratio model. すべての枝が異なる ω を持つ.
2: newick tree 内部に #1 のような印をつけることで,二つ以上の ω を推定する.
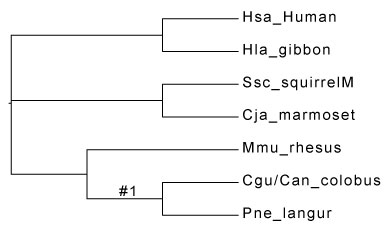
lysozymeSmall.trees ファイル: codeml.ctl で指定されている lysozymeSmall.trees ファイルを見てください.Newick format 内部に #1 を書き込むことで,ωを推定したい枝を選んでいます。それ以外は #0 としてプログラムは認識しています.FigTree で newic fromat を図にしてみましょう (以下)。
FigTree は「#」があると newcik format を読み込まないので,エディタで「#」を「@」などに変換して読み込ませてください.
ここでは,葉を食べるように適応したグループのリゾチーム (Yang 2009, P257) が,他のグループとは異なる ω を持つように設定されています.
*注意:newick format に枝長や BS 値,事後確率などが記入されていたら,エディタの検索置換機能を使って削除した方が良いです.
|
|
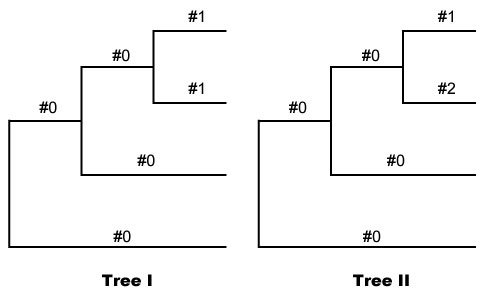
ちなみに,#1,#2 のようにいくつかの枝に異なる番号を指定すれば (#1 が複数の枝であっても良い),いくつかの枝に対して異なる ω が推定されるように設定されます (ずっと下の方にある、「二種類の ω を推定する場合」の図,Tree II を参照).
|
解析スタート: 以下のコマンドによって,codeml が自動的に codeml.ctl ファイルを読み込んで解析がスタートします.
./codeml
|
アウトプットファイル: 枝ごとに推定された ω が,mlcB2 という名前で保存したアウトプットファイルに newickformat としてまとめられています.
w ratios as labels for TreeView:
((Hsa_Human #0.6858 , Hla_gibbon #0.6858 ) #0.6858 , ((Cgu/Can_colobus #0.6858 , Pne_langur #0.6858 ) #3.5057 , Mmu_rhesus #0.6858 ) #0.6858 , (Ssc_squirrelM #0.6858 , Cja_marmoset #0.6858 ) #0.6858 );
その上のテーブルにも,枝ごとの ω がまとめられています.dS 値が極端に小さいか 0 の場合は,999 という値になってしまうようです.FigTree で図にしてみました (以下、右)。 |
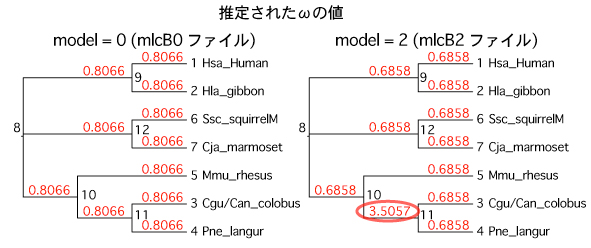 |
解釈: このデータは極端な例なので,ω が 3.5 などと大きな値が得られています.しかし,通常の解析では,ω が 1 以上になることは少ないようです.これは,ポジティブセレクションを受けているサイトはそのタンパク質の一部で,他のサイトは 純化選択 (purifying selection) を受けているため,配列全長を平均すると,1 よりも小さくなることが多いためです.
なお,ω がバックグラウンド (他の枝) よりも大きくなっている場合,2 つの解釈が考えられます (Yang 2009, P249).
A. どこかのサイトに正の自然選択が働いている.
B. 遺伝子全体の制約が緩くなって,淘汰圧の緩和 (リラクゼーション) が起きている.
B についてさらに詳しく考えると,有効集団サイズが小さくなることで,遺伝的浮動 (genetic drift) によって,偶然にアミノ酸置換が集団に固定しているために,ω が 1 に近くなっている可能性が考えられます.実際には B の場合,淘汰圧の緩和なのか,集団サイズの縮小 (e.g. ボトルネック) なのかは区別がつきにくいようです.この場合,いろいろな遺伝子を調べて,同じように緩和が見られれば,集団サイズの縮小によって ω が 1 に近くなっていることを示唆するようです.
尤度比検定: 検出された dN/dS の値が有意か統計的に検定します (詳しくは Yang 2009, P217).model = 0 の解析を行って,帰無仮説を検証します.model = 0 の計算は,すべての枝が同じ ω を持つと仮定します.この場合,model = 2 の計算よりもパラメータ数が 1 つ少ないので,model =0 と model =2 は包含 (入れ子状 [nested] の) 関係にあります.パラメータが少なくなるということは,より特殊は状況になっていることを意味するので,model = 0 は model = 2 の特殊な状態と言えます.
コントロールファイル (codeml.ctl) の該当する行を以下のように設定して,解析を行ってください.
outfile = mlcB0
model = 0
fix_omega = 0
計算が終了すると,アウトプットファイル (mlcB0) に以下が得られているはずです.
omega (dN/dS) = 0.80663
さらに,パラメータ数 (np: 13p) と対数尤度 (-906.017440) が表示されています.
TREE # 1: ((1, 2), ((3, 4), 5), (6, 7)); MP score: 65
lnL(ntime: 11 np: 13): -906.017440 +0.000000
np と対数尤度の値を使って尤度比検定を行います.カイ二乗値と自由度は以下の式で計算できます.
x^2 = 2*(ln modelA - ln model B)
d.f. = #p(modelA) - #p(modelB)
2*(ln modelA - ln model B) を、カイ二乗値の近似に利用しているようです (Hashiguchi et al. 2007, P173 左上 line 1)。
二つのファイルから得られた np と対数尤度の値をエクセル (CHISQ.DIST.RT) で計算します.下の図および lysozymeSmall/lnTEST.xlsx ファイルも参照してください.CHISQ.DIST.RT をエクセルで操作するには,下の図で fx と書かれた部分をダブルクリックしてください (エクセルのバージョンによっては,他の操作です).下の図,エクセルでは,A と B にそれぞれ model 2 と 0 で得られた尤度を書いています.CHISQ.DIST.RT の式そのままではダメなので,「2*」と,自由度の差である「1」を入れて,「=CHISQ.DIST.RT(2*(A1-B1),1)」を計算させています. |
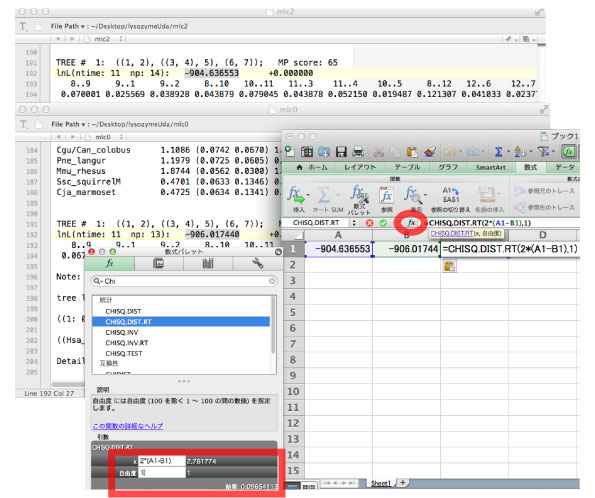 |
少し見えにくいですが,上の図でエクセルの左下,CHISQ.DIST.RT のコラム (濃い灰色) に,カイ二乗値 2.76, P value 0.097 が得られています.得られた P 値は,model=2 と model =0 の差異が,5% の水準では有意な差ではないことを意味しています.
|
二種類の ω を推定する場合
下図 II のように,#1 と #2 を tree file で設定してください.この場合,対立仮説と帰無仮説を model 2 で計算させて,包含関係として比較します.このように model の選択は,その都度 0 にするか 2 にするか考える必要があります. |
|
|
サイトモデルは,枝間の ω を変化させず,サイト間での ω の変異を検定するモデルです (Yang 2009, P261 参照).コントロールファイルを以下の設定にしてください.
outfile = mlcS
model = 0
NSsites = 0 1 2
NSsites を 0 1 2 にすると,帰無仮説も同時に計算します.
それぞれ,
Model 0: ωが均質.puryfying.
Model 1: puryfying か neutral という 2 つのカテゴリ.
Model 2: puryfying, neutral, positive という 3 つのカテゴリ.
を意味します.ここで言う「Model (マニュアルでも model と表記されている)」は NSsites パラメータを変化させた場合の Model で,コントロールファイルにあるオプション model = 0 とは別のものです.NSsites には,他にも 6 とか 7 があるようですが,2 が最も一般的なモデルです.前述のように,1 は 2 の特殊なケースと見なすことができます.0 はさらに特殊なケースと見なすことができ,neutral なサイトがないと仮定しています.計算を速くするテクニックとして,method という項目を調節すると良いそうです.
尤度比検定: mlcS に出力される自由度と対数尤度を用いてエクセルで尤度比検定を行います.Model 2 vs Model 1 あるいは Model 1 vs Model 0 という比較は意味があるそうですが,Model 2 vs Model 0 という比較は意味が無いそうです.
Model 0: one-ratio
TREE # 1: ((1, 2), ((3, 4), 5), (6, 7)); MP score: 65
lnL(ntime: 11 np: 13): -906.017440 +0.000000
...
omega (dN/dS) = 0.80663
...
Model 1: NearlyNeutral (2 categories)
TREE # 1: ((1, 2), ((3, 4), 5), (6, 7)); MP score: 65
lnL(ntime: 11 np: 14): -902.503872 +0.000000
dN/dS (w) for site classes (K=2)
p: 0.41271 0.58729
w: 0.00000 1.00000
...
Model 2: PositiveSelection (3 categories)
TREE # 1: ((1, 2), ((3, 4), 5), (6, 7)); MP score: 65
lnL(ntime: 11 np: 16): -899.998568 +0.000000
正の自然選択が働いているサイト: アウトファイルの最後の方に NEB および BEB 法として出力されます (Yang 2009, P264 を参照).
|
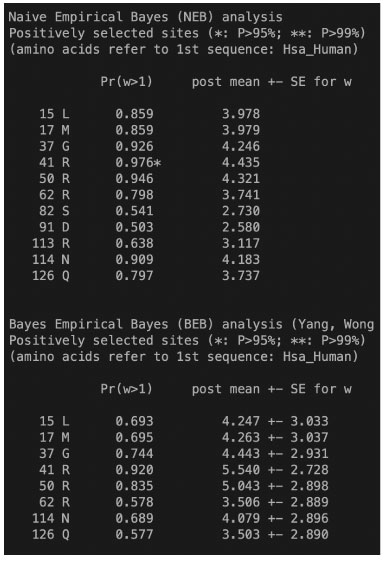 |
* は 95%, ** は 99% の有意水準のサポートを意味します.Pr は事後確率で,そのサイトに正の自然選択が働いている確率, post mean がそのサイトの ω 値をそれぞれ示します.作者の Yang さんは,マニュアルで BEB 法を薦めており,NEB は無視して良いと繰り返し述べています.
|
| 枝-サイトモデル (branch-site model) |
|
枝-サイトモデルは,枝モデルとサイトモデルをあわせた解析です (Yang 2009, P268 参照).ある特定の系統のあるサイトに働いている正の自然選択を検証するのに使います.枝モデルとサイトモデルは,それぞれすべてのサイトおよびすべての枝にわたって ω を平均するため保守的な結果しか得られないそうです.このため,枝-サイトモデルの方が検出力が高いそうです.
forground (#1: 興味のある枝) と background (#0: それ以外の枝) にわけて解析します.二種類の ω を推定する場合、図: Tree I のように,#1 か #0 という 2 種類の ω だけを設定した tree の解析しかできません.サイトが以下 4 つ (0, 1, 2a, 2b) の Site class に分けられます .background は帰無仮説なので ω2 ≥ 1 がないことに注意してください. |
| |
0 |
1 |
2a |
2b |
| 割合 |
p0 |
p1 |
(1 –p0– p1) p0/(p0 + p1) |
(1 –p0– p1) p1/(p0 + p1) |
| #0: background |
0 <ω0 <1 |
ω1 = 1 |
0 <ω0 <1 |
ω1 = 1 |
| #1: foreground |
0 <ω0 <1 |
ω1 = 1 |
ω2 ≥ 1 |
ω2 ≥ 1 |
0 <ω0 <1: 保存サイト (Negative).
ω1 = 1: 中立サイト (Neutral).
ω2 ≥ 1: 正の選択を受けているサイト (Positive).
この表は,マニュアル P31, Table 3 New model A を codeml アウトプット (下の図) に合わせて構成し直しました (Yang, 2009 P268 表 8.4 も同じ).
|
コントロールファイルで,
outfile = mlcBSwE
model = 2
NSsite = 2
fix_omega = 0
と設定して解析してください.
アウトファイル (mlcBSwE ファイル): 以下の箇所を探してください.
|
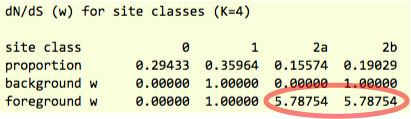 |
Site class 2b であれば, foreground が positive, background が neutral であるサイトの割合が 19% であることを示します.Site class 0 のサイトは 0 になっているので,強い puryfying selection が働いていることを示します.2a と 2b は foreground に正の自然選択が働いており,それぞれ ω が 5 以上と強い正の自然選択が働いていることを示しています.
尤度比検定: Site class 2a の ω,5.78754 を例として尤度比検定します.この場合は,Site class 2a と 2b の ω が 1 であることを仮定したモデルを帰無仮説として計算し、比較に使います.
まず先ほど解析で得られた結果が記されている mlcBSwE ファイルの以下の部分から,自由度と尤度を得ます.
TREE # 1: ((1, 2), ((3, 4), 5), (6, 7)); MP score: 65
lnL(ntime: 11 np: 16): -901.562791 +0.000000
次に,コントロールファイルで
outfile = mlcBSw1
fix_omega = 1
omega = 1
と設定して帰無仮説のもとで計算を行い,尤度とパラメータを得ます.
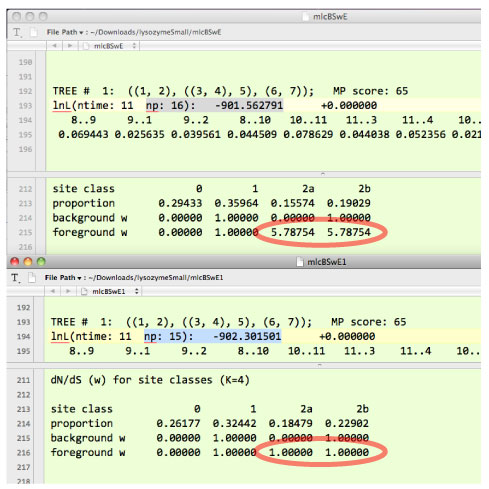
アウトファイル (mlcBSwE1 ファイル): 以下の箇所を探してください. 自由度とパラメータを mlcBSwE ファイルのものと比較して,尤度比検定を行います.
|
TREE # 1: ((1, 2), ((3, 4), 5), (6, 7)); MP score: 65
lnL(ntime: 11 np: 15): -902.301501 +0.000000
|
|
|
ガンマ補正は使えない
PAML の dN/dS 検定では,サイト間の進化速度不均質性を補正するガンマ補正が使えません.恐らく,ガンマ補正を行うことで計算速度が遅くなることを防ぐためです.このため,
fix_alpha = 1
を選択してください.
aaDist は使えない
アミノ酸の distance に関わるパラメータだそうです.どのアミノ酸も物理化学的に同じ距離と仮定した,簡単なモデルしか使えません.これも恐らく計算が複雑になることを防ぐためのようです.以下を選択してください.
aaDist = 0
アミノ酸組成は観察値
最尤推定していないそうです.観察値も一つのパラメータと見なすことができるようです.
|
|
論文はこちらを参照してください。
treeSAAP
dN/dS 検定を行うソフトウェアだそうです.
Introduction to calculating dN/dS ratios with codeml V.2
コマンドとともに、例題が gitHub から配布されています。
(1) アミノ酸配列をアライメント。
(2) pal2nal でアミノ酸アライメントに DNA アライメントを合わせる。
(3) codeml で枝モデル (NSsites = 0) によってペアワイス解析
(4) ダウンロードされた python script によって、結果を抽出。
|
|
|