|
|||
|
MacClade は,系統解析に用いる塩基 (アミノ酸) 配列をエディティングするソフトウェアです.祖先配列の推定や tree search などの機能もありますが,保存形式の変換や手動アライメント,topology 作成などの使い勝手の良さと美しさに関しては,MacClade の右に出るソフトウェアは無いと思います.さらに PAUP と互換性が良いことでも有名です.私の場合は,系統解析に際しては MacClade を使用している時間が最も長いと思います. アライメントファイルは基本的に MacClade で保存しておくと良いです.MacClade は手動でアライメントを行いやすいうえに,NBRF 形式で保存すれば,ドラッグ & ドロップで自動的に concatenate してくれます (最近バージョンアップされて,Fast や GenBank 形式も同様の操作が可能になりました).この場合 OTU の名前を統一しておく必要があります. |
|||
|
|||
|
MacClade を立ち上げると,ファイルは一つしか開くことができません.しかしアプリケーションを duplicate すれば,アプリケーションごとにファイルを開くことで,複数のファイルを同時に編集することができます.
|
|||
|
|||
|
以下の PAUP block を Sequence block の下にを設定し,PAUP で読み込んで execute します.「PHYLIP」を「NEXUS」に変更すれば,nexus 形式でも出力できます.
塩基配列データで 3rd codon を外したい場合は,Exclude で 3rd_codon を指定します.
|
|||
|
|||
|
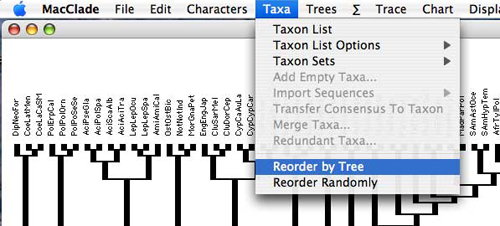
あらかじめ並べたい順序になっている Tree block を作成しておいて,ファイルを開きます.コマンド + T で tree モードにして,Taxa -> Reordered by Tree を選択してください.これを保存します.
|
|||
|
|||
| コマンド A で全配列を選んでください. Utilities から Complement を選び,その後 Reverse を選びます. |
|||
| Amino acid color |
|||
|
|
|||