|
|
|
|
BLAST+ は,類似配列検索プログラム集です.従来の local BLAST (legacy blast と呼ばれる) を発展させたものです.Legacy blast はさらなる開発とメンテナンスを中止したので,BLAST+ を用いた方が良いです.マニュアルはこちらです.
まずは,統合TV をご覧下さい.legacy blast と blast+ 間のオプション表があります.これまで local Blast (legacy blast)を使っていた方は,legacy blast と blast+ の対応表が役に立ちます.
データベース化には、SQLite (2.15.0以降) か LMDB (2.7.1以降) が使われています (link)。
|
|
[jun-inoue:inoue]$ ftp ftp.ncbi.nlm.nih.gov
Connected to ftp.wip.ncbi.nlm.nih.gov.
220-
Warning Notice!
You are accessing a U.S. Government information system which includes this
....
KERBEROS_V4 rejected as an authentication type
Name (ftp.ncbi.nlm.nih.gov:jun-inoue): anonymous
331 Anonymous login ok, send your complete email address as your password
Password:[email address でも良いですが,単にリターンでも大丈夫です]
230 Anonymous access granted, restrictions apply
....
226 Transfer complete
ftp> cd blast
250 CWD command successful
ftp> ls
227 Entering Passive Mode (130,14,250,12,196,27).
150 Opening ASCII mode data connection for file list
-r--r--r-- 1 ftp anonymous 20087 May 10 2004 blastftp.txt
dr-xr-xr-x 3 ftp anonymous 32768 Jul 9 01:10 db
dr-xr-xr-x 5 ftp anonymous 4096 Feb 27 13:56 demo
dr-xr-xr-x 6 ftp anonymous 4096 Apr 19 18:49 documents
dr-xr-xr-x 10 ftp anonymous 4096 Sep 14 2012 executables
dr-xr-xr-x 2 ftp anonymous 4096 Sep 11 2012 matrices
dr-xr-xr-x 7 ftp anonymous 4096 May 9 12:06 temp
dr-xr-xr-x 2 ftp anonymous 4096 Oct 7 2011 web_services
dr-xr-xr-x 2 ftp anonymous 4096 May 24 2012 windowmasker_files
226 Transfer complete
ftp> cd executables
250 CWD command successful
ftp> cd blast+
250 CWD command successful
ftp> cd LATEST
250 CWD command successful
ftp> get ncbi-blast-2.2.28+-universal-macosx.tar.gz
local: ncbi-blast-2.2.28+-universal-macosx.tar.gz remote: ncbi-blast-2.2.28+-universal-macosx.tar.gz
227 Entering Passive Mode (130,14,250,12,197,60).
150 Opening BINARY mode data connection for ncbi-blast-2.2.28+-universal-macosx.tar.gz (272477554 bytes)
226 Transfer complete
272477554 bytes received in 42 seconds (6.3e+03 Kbytes/s)
ftp> quit
|
|
ソースコードを使わない方法
インストールは単純で,ただダウンロードして得られた .pkg をダブルクリックするだけです.すでにコンパイルされたファイルが ncbi-blast-x.x.x+/bin ディレクトリに入っています (2018 年 3 月).
ダウンロードファイルに .pkg がない場合もあるようです.この場合は,おそらく bin ディレクトリにコンパイル済みの各種プログラムが入っています.これをコピー&ペースとして使ってください (2015 年 1 月).
ソースコードをコンパイル?
ソースコードからコンパイルも可能のようですが、blastp などは作成されず、以下のようなプログラムが作成されました。
https://www.ncbi.nlm.nih.gov/books/NBK569861/
jinoue@E73F3:~/ncbi-blast-2.15.0+-src/c++/ReleaseMT/bin$ ls
cleanup-blastdb-volumes.py lmdb_test2 project_tree_builder
datatool lmdb_test3 run_with_lock
get_species_taxids.sh lmdb_test4 test_pcre
legacy_blast.pl lmdb_test5 update_blastdb.pl
lmdb_test1 lmdbxx_sample windowmasker_2.2.22_adapter.py
ncbi-blast-2.15.0+-src.tar.gz をダウンロードし、以下を行いました。
cd ncbi-blast-2.15.0+-src/c++
./configure
make
sudo make install
./configure は数秒で終わりましたが、make は 1 時間ぐらいかかりました (2024 年 9 月)。
基本的なテクニック
余談ですが, 基本的なテクニックを紹介します。
以下を入力すると,インストールされたプログラムの場所がわかります.
which makeblastdb
cd はディレクトリに入るコマンドです。以下では、/usr/local/ncbi/blast/bin に入ります。
cd /usr/local/ncbi/blast/bin
open はディレクトリをウィンドウとして開くコマンドです。/usr/local/ncbi/blast/bin がウィンドウとして開きます.
open /usr/local/ncbi/blast/bin
|
| データベースの作成,blastp 検索,配列の取得 |
|
blastPlusExample.tar.gz 使って説明します.Quick start を参照しました (2019 年 7 月作成、2024 年 10 月改訂).
以下をターミナルから入力してください.
sh 000command.sh
以下,000command.sh の解説です.
# A ) アミノ酸配列データの解析。
#
prot データベースを作成します。
./makeblastdb -in db_prot.txt -dbtype prot -parse_seqids
# -parse_seqids:
seq ID 検索を可能にします.
# blastp 検索を行います.
./blastp -query query_prot.txt -db db_prot.txt -num_alignments 10 -evalue 1e-12 -out out_protREC.txt
# seq ID を用いて「指定したデータベース,レコード,範囲」の配列を取得します.
./blastdbcmd -entry ENSP00000362111.4 -db db_prot.txt -dbtype prot -out out_protSEC.txt
# B) 塩基配列データの解析。
#
nucl データベースを作成します.
./makeblastdb -in db_nucl.txt -dbtype nucl -parse_seqids
# blastn 検索を行います.
./blastn -query query_nucl.txt -db db_nucl.txt -num_alignments 10 -evalue 1e-12 -out out_nuclREC.txt
# seq ID を用いて配列を取得します.
./blastdbcmd -entry ENST00000373020.8 -db db_nucl.txt -dbtype nucl -range 100-150 -out out_nuclSEC.txt
*makeblastdb で作成されたデータベースは,legacy Blast (blastall) でも使うことができました.また,逆も可能で,formatdb (legacy Blast) で作成されたデータベースを blastp (blast+) で使えました.しかし,一応 makeblastdb によるデータベース化は、blastn や blastp のバージョンに合わせて毎回やり直しています.
|
| なお Mac では、「“blastp”は、開発元を検証できないため開けません。」というエラーメッセージが出ます。このページの下の方に「Mac: プログラムの「開発元を検証できない」と言われた場合」を参照して対応してください。 |
 |
|
| |
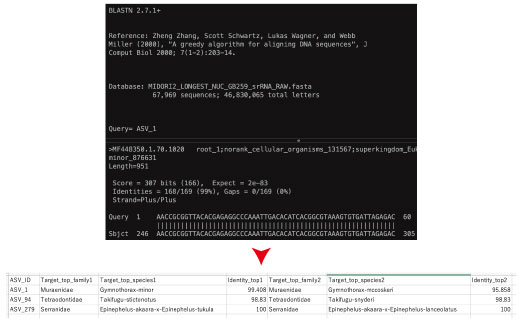
| blastn results から .csv ファイルを作成 |
|
Qiime2 のページ、「BLAST+ を用いた種判定」を参照。 |
 |
| (2024 年 3 月) |
| |
|
各種パラメーターは,以下のコマンドで確認できます.
$ blastn -help
- task (Manual, P7 "BLAST+ features: Tasks")
塩基配列の検索には,megablast (種内解析用) と blastn (種間解析用) が存在する.blastn はデフォルトだと megablast が選ばれる.
- outfmt (Manual, P33)
アウトファイルの形式を設定する.
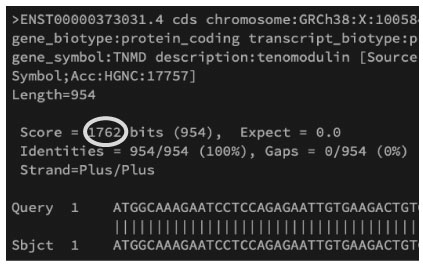
- bit score (Help)
evalue はデータベースのサイズに依存するので,bit score を使った方が良いと言う意見もある. |
|
- parse_seqids
makeblastdb で利用可能なオプション。ID 検索を可能にするデータベースを作成できる。Manual によると、
Parse bar delimited sequence identifiers (e.g., gi|129295) in FASTA input.
とあり、データベース化する fasta name line を ref|129295| のように ID を bar で囲んでおく必要がある。
|
| fasta file から配列を取得する: blastdbcmd |
|
| blastdbcmd によって,position 情報 (コーディネートとも言う) を元に,fasta file から配列を取得します.extractSeqFromFasta.tar.gz 使って説明します. |
# -parse_seqids オプションを用いて,blastdbcmd で利用可能なデータベースを作成します.
$ makeblastdb -in test.fa -dbtype nucl -parse_seqids
Building a new DB, current time: 08/30/2019 12:02:51
...
# 作成したデータベースから,blastdbcmd によって目的の配列を取得します.
$
blastdbcmd -db test.fa -dbtype nucl -entry ENST00000373020.8 -range 5-10
>ENST00000373020.8:5-10
CGTCCC
$ cat test.fa
>ENST00000373020.8
ATGGCGTCCCCGTCTCGGAGACTGCAGACTAAACCAGTCATTACTTGTTT
blastdbcmd については,NCBI の公式マニュアルを見ても,あまりよい説明を見つけられませんでした.「blastdbcmd -help」によってオプションを確認してください.おそらく, bedtools getfasta (以下のコラム) のように,repeat 配列を小文字のまま表示するオプションはありません.(2019 年 8 月).
BEDTools の getfasta を用いても,fasta file から配列を得られます.こちらを参照してください.
|
| 種ごとにまとめられた ftp サイトのデータをまとめてダウンロード |
|
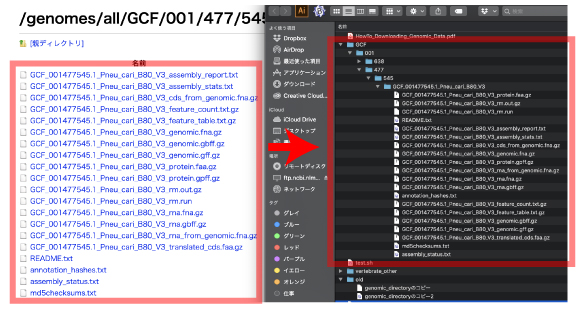 |
BLAST Help, PDF にある HowTo_Downloading_Genomic_Data.pdf, P44,にしたがっています.Case 2 にある方法です.
ftp アドレスを用いてデータをダウンロードする
genomic_directory に以下の 2 行を保存します.これらのアドレスは,NCBI の ftp サイトにWebブラウザから入って得ます.
ftp://ftp.ncbi.nlm.nih.gov/genomes/all/GCF/001/477/545/
GCF_001477545.1_Pneu_cari_B80_V3/
ftp://ftp.ncbi.nlm.nih.gov/genomes/all/GCF/001/638/985/
GCF_001638985.1_Phybl2/
以下のコマンドによって,genomic_directory に記述されている ftp ディレクトリに存在するファイルを一気にダウンロードできます.
wget -r --no-parent --no-host-directories --cut-dirs=2 --level=1 \
--input-file=genomic_directory
wget のオプションの説明は以下.
-r: recursively works through the directory
--no-parent: ignores the parent directory
--no-host-directories: saves the files without prepending the NCBI FTP URL
--cut-dirs=2: saves the files without creating intermediate directories
--level=1: works only at that level of directory
--input-file=value: sets directory input to the file specified by value
ある分類群のデータを一気に得る
fungi を例にしています.NCBI がまとめている fungi の ftp サイトでは, assembly_summary.txt にそれぞれの種の ftp アドレスがまとめられています.以下のコマンドによって,assembly_summary.txt から,これらのアドレスを全て genomic_directory というファイルに保存します.
curl 'ftp://ftp.ncbi.nlm.nih.gov/genomes/refseq/fungi/
assembly_summary.txt' | \
awk '{FS="\t"} !/^#/ {print $20"/"}' > genomic_directory
あとは,wget によって genomic_directory に保存した ftp アドレスのファイルをダウンロードします.
(2019 年 6 月)
|
|
アドレスにスペースがあるとエラーが出ることがある
makeblastdb2.16.0 のエラーメッセージです。2.12.0以前では、このエラーは出ませんでした。
BLAST Database error: Database memory map file error
ただ、データベース化は正常に進んだようで、その後行った blastn と blastdbcmd は、問題のない結果が得られませいた。
ディレクトリのアドレスにスペースが入っているとエラーが出てしまうそうです (link)。例えば、ホームディレクトリ (~) で解析すると、エラーは消えました 。以下、「スピード比較」を参照してください (2024 年 11 月)。
|
makeblast 2.10.0 以降?は nameline が 50 文字まで
makeblastdb 2.10.0 は、 データベースにする fasta file の name lineが 50 文字以上だと動かないです。
BLAST Database creation error: Near line 9643, the local id is too long. Its length is 58 but the maximum allowed local id length is 50. Please find and correct all local ids that are too long.
makeblast 2.16.0 でも同様のメッセージを確認しました。
なお、-parse_seqids オプションを外すとエラーは消えます。
(2024 年 10 月)。
|
makeblast: 古いバージョンのデータベースは利用できる
あまりお勧めできませんが、古いバージョンの makeblastdb で作成されたデータベースを、新しいバージョンの blastn で利用できます。例えば、makeblastdb 2.7.1 は正常に作動し、作成されたデータベースは blastn2.16.0 で解析できます。しかし、逆、つまりmakeblastb2.16.0 で作成されたデータベースは、blastp2.7.1 では解析できないです。
(2024 年 10 月)。
|
スピード比較
blastdbcmd_speedtest.tar.gz (パスワードが必要です)
makeblastdb, blastn, blastdbcmd で比較しました。
2.11.0 以降は遅い:
およそ、makeblastdb は 2 倍、blastn は 7 倍、blastdbcmd は 20 倍、遅くなっています。
| |
elapsed time |
| バージョン |
makeblastdb |
blastn |
bladtdbcmd |
| 2.7.1 |
0.0228 |
0.0025 |
0.0009 |
| 2.10.1 |
0.0228 |
0.0035 |
0.0009 |
| 2.11.0 |
0.0411 |
0.0195 |
0.0171 |
| 2.12.0 |
0.0404 |
0.0205 |
0.0186 |
| 2.16.0 |
0.0392 |
0.0210 |
0.0205 |
| |
2.11 以降遅い |
2.11 以降遅い |
2.11 以降遅い |
解析 2 回目以降に速くなる:バージョンを変えて再解析しはじめた場合、最初よりも 2 回目以降の方が、格段にスピードが速いです。これは、makeblastdb, blastn, blastdbcmd に共通して見られる現象です (blastp も同様の経験があります)。そこで、上記の表では、数回解析して安定した数値を使っています。 当初は、makeblastdb によるデータベース化直後は、blastn と blastdbcmd などで検索が遅くなると思っていました。しかし、makeblastdb 自体も 2 回目以降が速くなっているし、バージョン変更後に makeblastdb を毎回やり直しても blastn など検索のスピードは 2 回目以降で速くなりました。つまり、データベース化されて 2 回目以降、blastn などが速くなっているわけではなさそうです。
makeblastdb が産出する .n** ファイル:
| makeblastdb -dbtype nucl -parse_seqids 産物 |
| |
.nto |
.ndb |
.nhr |
.nin |
.njs |
.nog |
.nsd |
.nsi |
.nos |
.not |
.nsq |
.ntf |
.nto |
| 2.7.1 |
- |
- |
+ |
+ |
- |
+ |
+ |
+ |
- |
- |
+ |
- |
- |
| 2.10.1 |
- |
+ |
+ |
+ |
- |
+ |
- |
- |
+ |
+ |
+ |
+ |
+ |
| 2.11.0 |
- |
+ |
+ |
+ |
- |
+ |
- |
- |
+ |
+ |
+ |
+ |
+ |
| 2.12.0 |
- |
+ |
+ |
+ |
- |
+ |
- |
- |
+ |
+ |
+ |
+ |
+ |
| 2.16.0 |
+ |
+ |
+ |
+ |
+* |
+ |
- |
- |
+ |
+ |
+ |
+ |
- |
| *ディレクトリアドレスにスペースが入っていると、.njs ファイルは作成されません。エラーメッセージ "BLAST Database error: Database memory map file error" |
レコード数では、ほとんど変化しない:なお、データベースのレコード数が多くなった場合、blastn は若干遅くなりますが、blastdbcmd は変化しないです。
| |
elapsed time |
| レコード数 |
makeblastdb |
blastn |
blastdbcmd |
| 5 |
0.0207 |
0.0188 |
0.0180 |
| 106989 |
0.0404 |
0.0242 |
0.0188 |
| |
遅くなる |
少し遅い |
変化なし |
| 2.12.0 を使いました。 |
(2024 年 11 月)。
|
| |
|
| BLAST 結果のデータベース・アドレス |
おそらく、データベースが大きいと、結果にデータベースのファイル名だけが表示され、アドレスは表示されないです。
BLASTN 2.7.1+
Reference: Stephen F. Altschul, Thomas L. Madden, Alejandro A.
Schaffer, Jinghui Zhang, Zheng Zhang, Webb Miller, and David J.
Lipman (1997), "Gapped BLAST and PSI-BLAST: a new generation of
protein database search programs", Nucleic Acids Res. 25:3389-3402.
Database: LONGEST_srRNA_RAW.fasta
67,969 sequences; 46,830,065 total letters
####
BLASTN 2.7.1+
Reference: Stephen F. Altschul, Thomas L. Madden, Alejandro A.
Schaffer, Jinghui Zhang, Zheng Zhang, Webb Miller, and David J.
Lipman (1997), "Gapped BLAST and PSI-BLAST: a new generation of
protein database search programs", Nucleic Acids Res. 25:3389-3402.
Database: ./PMifish_Results/Scorpaenodes-evides_7/004_userDB.txt
15,176 sequences; 2,600,463 total letters
(2024 年 10 月)
|
|
| Mac: プログラムの「開発元を検証できない」と言われた場合 |
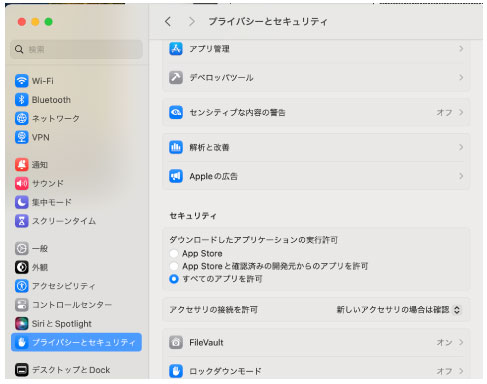
| 解決方法 1: すべてのアプリを許可する |
システム設定 > プラバシーとセキュリティ > すべてのアプリを許可 |
 |
macOS Sierra 以降では、「全てのアプリを許可」が削除されているそうです。表示させるためには、terminal で以下を入力します。
$ sudo spctl --master-disable
元に戻したい場合は、以下です
$ sudo spctl --master-enable
こちらを参照しました。
(2024 年 11 月)
|
| 解決方法 2: 毎回変更する |
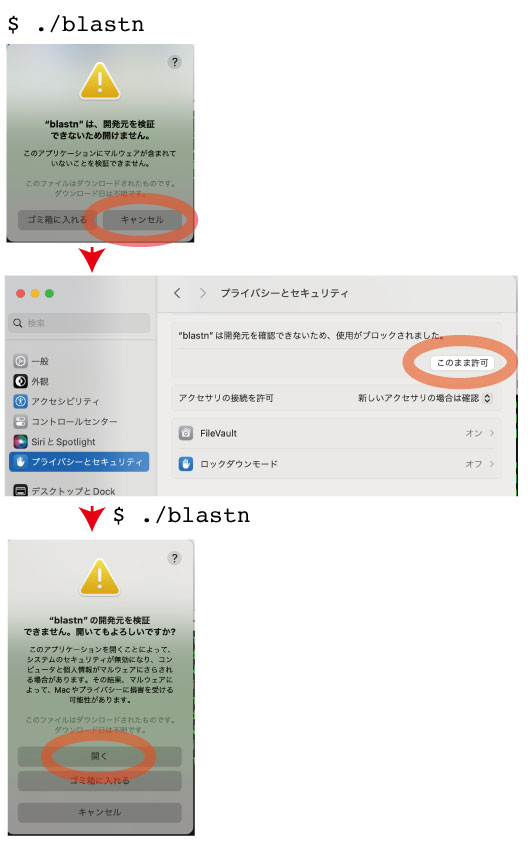
(2024 年 3 月) |
| |
|
BLAST+ 公式ページ
最近のマニュアル (PDF ヴァージョンもある) が得られる.
BLAST Help, PDF
PDF マニュアル.種ごとのゲノムデータダウンローについてはこちら (HowTo_Downloading_Genomic_Data.pdf, P44).
NAGRP BLAST Server Documentation
Mega blast などについて、BLAST+ 本家のページより丁寧な解説があります。
PSI-BLAST
統合テレビ。NCBI website。部分特異的スコア行列 (PSSM: Position Specific Scoring Matrix) を利用して、類縁の配列を探す方法の紹介。
mac でバイオインフォマティクス
NCBI からバクテリアゲノムをダウンロードする
mac でバイオインフォマティクス
nr などの NCBI データベースをダウンロードする ncbi-blast-dbs
fastacmd の後継者:blastdbcmd
ゲノム配列の任意の場所を切り出すソフトウェアの紹介.
BLAST FASTA Files Will No Longer Be Available on the FTP Site Effective April 2024
fasta が利用できない?
|
|





