|
|
|
|
このページは古い情報が書かれています.最近は BEAST 2 が出ているので,そちらを使った方が良いようです (2016 年 7 月).
|
BEAST はベイズ法に基づいて系統樹探索と分岐年代推定を行うソフトウェアです.さらに,coalescent theory に基づいた集団解析 (過去の有効集団サイズの推定など) も行えます.ここでは年代推定のやり方を解説しますが,集団解析も手順はかなり似ています (2016 年 12 月).
1) input file の作成 (BEAUti),2) 実際の解析 (BEAST),3) 結果の解釈 (Tracer),3) tree の描画 (FigTree),といくつかのプログラムに役割分担がなされています.Mac, Windows, Unix, Java に対応しているため,だいたいの PC で解析を行えます.2007 年になってマニュアルと HP が整備され始めました.
multidistribute と比較すると,樹形探索と年代推定を同時に行い,進化速度を枝ごとに関連せずに推定する,という点が大きな違いです.特に後者は,multidistribute のように進化速度を枝間で関連させて (autocorrelation) 推定すると,速度の急激な変化を検出しにくい,と考えられているようです.他にも,各種パラメーターの事前確率に異なる分布を選べる点が特徴としてあげられます.
おおまかには言うと,nexus ファイルを BEAUti で読み込んで,各種の設定を行い, .xml ファイルとして保存して BEAST で走らせます.BEAUti ではまだ対応しきれない設定が多く,これらはエディタを用いて .xml ファイルを直接書き直すことになります.うまく動くと,解析の進み具合を示す棒グラフが表れます.問題がある場合は,棒グラフが出る前にエラーメッセージが出ます.
注意 -1 : 私は BEAST を使いこなしてはいないので,論文に使うときは解析方法などより詳細に調べた方が良いです [2010 年 10 月].論文には使いませんでしたが,Inoue et al. (2009) のデータで作成した例題を作りました [2011 年 1 月].
注意 -2 : 最近はかなり使いやすくなったという噂を聞きました.このウェブサイトの情報はかなり古い可能性があります.マニュアルなどをまず参照してください (2016 年 2 月).
|
|
|
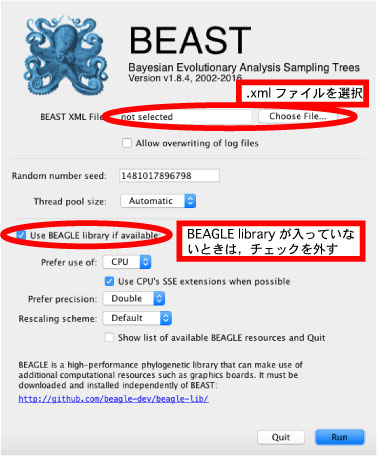
- BEAGLE というライブラリを使うと解析がかなりスピードアップされるようです.こちらからダウンロードしてください (2016 年 12 月).
|
|
|
- Tutorials が充実してきました [2011 年 1 月].
- Idaho で行われた Evolution 2009 に参加してきました.ほとんどが BEAST を用いて年代推定の解析を行っていました.ただ,BEAST はマニュアルがないので,研究グループ内で情報を交換して解析を進めているそうです.BEAST で解析を行うには,BEAST を使いこなしている人が周辺にいて,詳細な点を教えてもらう必要があるように思えます [2009 年 6 月].
- Time constraint の prior distribution に関する説明はこちらが参考に成ります.
- Hedges and Kumar (2004) in Trends in Genetics
- Benton and Donoghue (2007) in Molecular Biology and Evolution
- Ho (2007) in Journal of Avian Biology
- アミノ酸組成は mcmc サンプリングによってベイズ推定しているわけではないみたいです (アミノ酸の解析ではアミノ酸組成を考慮した F モデルはまだ使用できないみたいです).作成した .xml ファイルをエディターで見てみると,hkyModel id="hky1" などで frequency を設定しているのに,最後の方にある operators では kappa があるのに frequency がありません.疑問に思っていたら,こちらのページの真ん中ぐらいに
Note that, in all 4 substitution models, the base frequencies are set empirically from the whole alignment and although each partition has a frequencies parameter, these are not estimated in the MCMC (although they could be).
という記述を見つけました.おそらく observed (それぞれの OTU の平均値)
を解析に用いていると思います [2007 年].
- とりあえず動かすには,なるべく default のままで解析を行った方が良いようです.分岐の制約以外の prior を変更しないようにしたら,なんとか解析が動き出しました.MCMC の generation は,動作確認に際して 10000 回にしても問題なく動きました [2007 年].
- Unix バージョンは PC によっては動かない場合があるようです.
コンパイルも不要 (?) なようです.Unix バージョンといっても,実際は bin/beast にシェルスクリプトが書いてあって,lib/beast.jar を走らせるしくみになっています [2007 年].
- アミノ酸の解析も塩基配列と同様の手順で行うことができます.partition も 13 個にして問題なく動きました.アミノ酸の場合は rate matrix を推定しないので,partition の設定が若干異なります.また,F 補正 (データから得られるアミノ酸組成値を解析に反映させる) には対応していないようです [2007 年].
- HP などを見ていると,作者らが user に分かり易く説明するよう心がけていることがわかります.しかし,私は先日ようやく一通りの結果を出せるようになったにすぎませんが,残念ながらまだエラーも見られ,マニュアルや HP だけでは理解できない部分があると思いました [2007 年].
|
|
|
Nexus 形式で作成しておき,BEAUti で読み込みます.PAUP や MacClade で作成されたブロックは読み込まないので,エディターで削って sequence だけの最もシンプルな形にしておきます.partition の設定は最後にエディター上で手動で行うことになります.
|
|
|
- 年代推定に際して制約を比較的多く設定する場合は,user specified の starting tree を設定する必要があります.制約が少ない場合は,starting tree を UPGMA にしても動きます.
- 制約をたくさんの node に施す場合は,starting tree を使わざるを得ないと思います.starting tree は樹長付きである必要があります.このため,私は multidistribute で得られた時間軸付き系統樹を starting tree として用いています.RAxML で得られた ML tree でも動きました.
- starting tree は最後にエディターを用いて .xml ファイルに書き込みます (後述).こちらのサイトによると以下のように設定すると,starting tree が fix されて使われるようです.いずれにしても BEAUti で設定は不可能なので,のちほど xml ファイルをエディターで開いて切り貼りすることになるようです [2011 年 1 月].
変更前
<!-- Generate a random starting tree under the coalescent process -->
<coalescentTree id="startingTree">
<constrainedTaxa>
<taxa idref="taxa"/>
<tmrca monophyletic="true">
<taxa idref="ingroup"/>
</tmrca>
</constrainedTaxa>
<constantSize idref="initialDemo"/>
</coalescentTree>.
変更後
<newick id="startingTree">
(tree in newick format);
</newick>
- monophyly を仮定してその根幹の分岐に年代の制約を施す場合は,BEAST の解析が最初の段階で止まってしまうことがあります.これは,仮定した monophyly に対するデータの支持する確率が,generation のスタート時にあまりに低いために生じるようです.まだ確証は得られていませんが,分岐の年代制約を与えるときに,monophyly を選択しなければこの問題はクリアできるような気がします.
|
|
|
今のところ以下の条件で解析が走るようになりました.これから徐々に丁寧な記述に書き換えていきます.詳細はマニュアルと HP にある tutorial,Mailing List の質疑応答を参照して下さい.
Mac version で作動する簡単な例題を作成しました.v. 1.5.3 で動作確認しました.
こちらからダウンロードしてください.
この例題は,とりあえず一連の操作が短時間で作動するファイルであることを理解しておいてください.私は Beast の結果を論文として投稿したことがないので,不十分な点が含まれていると思います [2010 年 1 月].
BEAUti
- BEAUti を用いて nexus ファイルを読み込みます.Taxa タブと Priors タブのページを行き来して,いくつかの分岐の制約に対する制約を施します (Tutorial 2).
- codon position で partition を設定します.BEAUti ではコドン別にしか設定できませんが,エディターを用いて遺伝子別などの partition なども設定できます (後述).この際に置換モデルと rate heterogeneity model を partition で unlink にします.これらは後で xml ファイルをエディターで直して codon 意外の領域に対する設定も行います.
- Priors のページで,UPGMA tree を starting tree とする設定を行います.starting tree は後ほどエディターで書き換えることができます.
- .xml ファイルとして保存します.
*余談ですが,BEAUti と BEAT は美女と野獣 (Beauty and the beast) にかけているようです.
エディタ
- BBEdit などのエディタで xml ファイルを開き,UPGMA tree の設定を user defined の starting tree 設定に書き換えます (Tutorial 4).制作者に聞いたところ,starting tree は樹長付きでないと作動しないそうです.私は multidistribute の結果を starting tree として使っています (この場合,樹形探索に用いた外群を外して解析しています).
- partition を設定します (Tutorial 6).こちらのページも参考になります.
- 実際の推定値は,.xml ファイルの statistic にあたる部分に対象とする分岐の設定を加える必要があります. BEAUti で constraints を施した分岐は自動的に statistic が作られますが,他のノードに関してはエディタで手入力する必要があります (Tutorial 2)
BEAST
- 作成した .xml ファイルを BEAST で走らせます.最初のうちは,以上に示した ステップの途中で .xml ファイルを出力し,うまく作動するかどうか確認しながら作業を進めた方が良いと思います.慣れてくると,BEAUti で新たな設定を作成して保存してから必要な箇所をテキストとして取り出し,これを既存の .xml ファイルに継ぎ足して解析条件を変更することができます
LogCombiner (ダウンロードしたファイルに入っています)
- 別々に走らせた BEAST の log (あるいは tree) ファイルをまとめます.異なるデータやモデルによって得られた log ファイルを combine しても意味がないそうです.
- 割と時間がかかります.10,000,000 generation の解析を 別々に走らせて得られた log ファイルを一つにまとめたところ,私の Mac G4 では 5 分ぐらいでした.tree ファイルはもっと時間がかかりました.
- 得られた out ファイルをエディターで開けてみると,4 倍のデータ数と成っており,generation の番号も書き換えられていました.
Tracer
- .log ファイルを読み込んで解析します.burnin は自動的に 10 分の 1 にされますが,変更可能です.
- 数字としての分岐年代は,Tracer で解析して得られます.このため,特定の分岐に対する年代が必要な場合は,あらかじめ BEAUti の Taxa ページで分岐群としての設定して BEAST を走らせる必要があります.
- Analysis BEAST output が参考になります.
TreeAnnotator (ダウンロードしたファイルに入っています)
- .trees ファイルの解析を行います.burnin したうえで pooled trees の consensus を行い,時間軸付きの tree と事後確率を保存します (Nexus 形式になります).burnin は generation 数ではなく tree 数です.Node heights は Mean heights にしておいた方が良いと思います.
FigTree
- TreeAnnotator で保存したファイルを読み込み,tree を描画します.分岐年代の信頼区間も簡単に図にすることができます.
|
| Google earth に移動経路をマッピングする |
|
私はやったことがありませんが,地球規模の交雑や移動経路を Google earth 上にマッピングすることができるようです.図がかっこいいです.こちらのサイトをご覧下さい.
Lemey et al. (2009).
|
|
A Rough Guide to BEAST 1.4
こちらによると,もっともマニュアルに近いものだそうです.
BEAST Google Groups
質問コーナーです.私が質問したときは,わりと親切な答えが作者や他のユーザーから帰ってきました.作者の Drummond さんや Rambaut さんも返答してくれるようです.
|
|
|
Drummond AJ, Ho SYW, Phillips MJ, et al. 2006
Relaxed phylogenetics and dating with confidence. PLOS BIOLOGY 4 (5): 699-710 MAY 2006
BEAST がどのようなメカニズムで動いているか説明しています.
Drummond AJ & Bouckaert RR (2015)
Bayesian evolutionary analysis ; with BEAST (Cambridge University Press, Cambridge, United Kingdom) pp xii, 249 pages. Amazon.
BEAST 2 の解説書.
Nielsen, R & Slatkin, M. (2013)
An Introduction to Population Genetics: Theory and Applications. Sinauer Associates Inc. Amazon.
Coalescent theory に関する本.
Heath TA (2017) Divertence time estimation using BEAST v2. Link.
Beast 2 を用いた分岐年代推定のチュートリアル.図も多用しわかりやすそう.
|
|
|